Martín Femenias, becario doctoral en el IPEEC-CONICET en el Grupo de Herpetología Patagónica-LASIBIBE, publicó un interesante artículo en la revista Bioinformatics: ExplorATE: A new pipeline to explore active transposable elements from RNA-seq data.
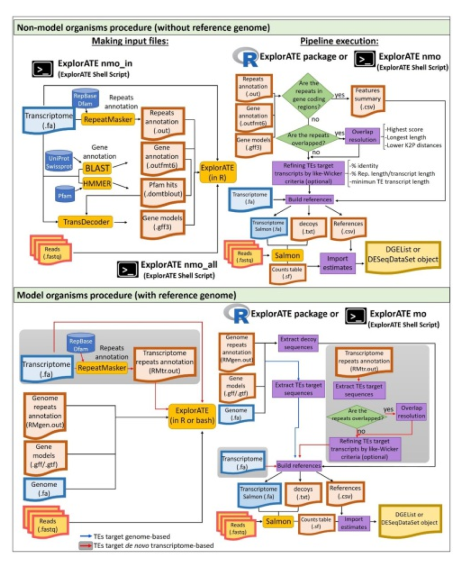
Los elementos transponibles (TE) son ubicuos en los genomas y muchos permanecen activos. Los TE comprenden una fracción importante de los transcriptomas con efectos potenciales sobre el genoma del huésped, ya sea generando mutaciones deletéreas o promoviendo novedades evolutivas. Sin embargo, su estudio funcional está limitado por la dificultad en su identificación y cuantificación, particularmente en organismos no modelo. Femenías et al. (2022) desarrollaron una nueva metodología (ExplorATE o Explore Active Transposable Elements) implementada en R y bash que permite la cuantificación de TE activos en organismos modelo y no modelo. ExplorATE crea índices específicos de TE y utiliza la alineación selectiva (SA) para filtrar los transposones cotranscriptos dentro de los genes en función de las puntuaciones de alineación. Además, el software incorpora criterios similares a Wicker para refinar un conjunto de TE de destino y evitar el mapeo falso. Con base en datos simulados y reales, se encontró que la estrategia SA adoptada por ExplorATE logró mejores estimaciones de elementos no cotranscriptos que otros softwares disponibles basados en alineación o mapeo. Los resultados de ExplorATE mostraron una alta congruencia con las herramientas basadas en la alineación con y sin un genoma de referencia, pero ExplorATE requirió menos tiempo de ejecución. Del mismo modo, ExplorATE amplía y complementa la mayoría de los análisis TE anteriores al incorporar los efectos de cotranscripción y mapeo múltiple durante la cuantificación, y proporciona una integración perfecta con otras herramientas posteriores dentro del entorno R. El código fuente está disponible en:
https://github.com/FemeniasM/ExplorATEproject and https://github.com/FemeniasM/ExplorATE_shell_script.
Supplementary information: Supplementary data are available at Bioinformatics online.